Proteínas: Niveles de organización
Estructura primaria
La secuencia de aminoácidos en una cadena polipeptídica determina su estructura primaria. Esta secuencia se especifica por la información genética. La primera proteína cuya secuencia se descubrió fue la insulina.
Estructura secundaria
Organización de la cadena polipeptídica en el espacio mantenida por puentes de hidrógeno entre los elementos del enlace peptídico (C=O y NH). Permite que la cadena adquiera dos tipos de estructura comúnmente: helicoidal (se enrolla como un cilindro alrededor de un eje) y laminar (plegada en forma de lámina). Se pueden encontrar en todos los tipos de proteínas. Las estructuras más estables y frecuentes son hélice α y hoja β (propuestas por Linus Carl Pauling y Elias James Corey).
Hélice α: hay 3,6 aminoácidos por cada vuelta, y cada vuelta tiene 5,4 Ángstrom. Todas las cadenas laterales de los aminoácidos se quedan hacia fuera. Es muy frecuente porque es muy estable y es siempre dextrógira. esta estructura está estabilizada por muchos puentes de hidrógeno (el mayor número posible), todos los grupos carbonilo forman puente de hidrógeno con el tercer aminoácido tras él. Los enlaces de hidrógeno son paralelos al eje de la hélice por lo que la molécula no puede ser estirada. Hay proteínas que son α-hélice al 100 % pero otras pueden presentar menor porcentaje o no presentar nada.
Aminoácidos incompatibles:
Hay aminoácidos incompatibles con esta estructura debido a las propiedades de su cadena lateral.
- El aspártico tiene la cadena lateral cargada por lo que si hay muchos juntos se repelen desestabilizando la molécula. Sólo son estables si no están disociados
- Hay aminoácidos que son muy voluminosos en su carbono β porque están sustituidos y el residuo no puede extenderse. Son la treonina y la isoleucina
- Hay otro aminoácido que rompe la α-hélice que es la prolina. Como el enlace no puede girar la cadena da la vuelta. Ya que tiene el grupo α-amino sustituido no puede formas puentes de hidrógeno. Se puede tener α-hélice antes y después de la prolina
Una proteína puede tener α-hélice a lo largo de un trozo porque los aminoácidos son compatibles. La cadena se puede estirar rompiendo puentes de hidrógeno y el límite está en la cadena polipeptídica.
Hoja β u hoja plegada:
Estructura secundaria mantenida por puentes de hidrógeno entre elementos de la cadena peptídica. Aspecto laminar.
Diferencias con α-hélice:
- La cadena polipeptídica está lo más extendida posible
- Distancia entre aminoácidos 3,5 ángstrom
- Estructura mantenida por puentes de hidrógeno, pero en lugar de formar puentes entre elementos de la misma cadena es con elementos de cadenas distintas (o de la misma que da la vuelta). Los puentes de hidrógeno se establecen perpendiculares al eje de la lámina. Todos los carbonilo forman puentes de hidrógeno con los amino, por lo que es igual de estable que la α-hélice. Cuando hay 2 trozos existen 2 posibilidades:
- Si los 2 segmentos tienen direcciones distintas la hoja plegada es antiparalela. como los puentes de hidrógeno son enlaces direccionales es más estable
- Si los 2 segmentos tienen direcciones iguales la hoja plegada es paralela
Las cadenas laterales van por encima y por debajo alternativamente porque la configuración del enlace es trans.
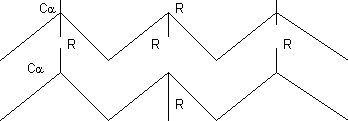
Las cadenas laterales están más próximas que en la α-hélice, por lo que sólo es compatible con aminoácidos de cadena lateral muy corta: glicina, alanina y serina (son los más pequeños). Los otros aminoácidos sufren repulsiones de tipo estérico. En realidad como la cadena está un poco retorcida cadenas más grandes caben.
Giro β:
Como la proteína se pliega sobre sí misma ha de cambiar de dirección y ser estable. De todos los giros éste es el más estable y frecuente. Es un elemento de estructura secundaria porque está estabilizado por puentes de hidrógeno entre el carbonilo y el amino del enlace peptídico. Está formado por 4 aminoácidos y estabilizado por un puente de hidrógeno formado entre el carbonilo del primer aminoácido y el amino del cuarto. Como hay poco espacio uno de los aminoácidos será pequeño, el tercero suele ser glicina y otro prolina, que tiene problemas de tipo estérico pero es el que se necesita para cambiar la dirección.
Estructura supersecundaria
Asociaciones entre estructuras secundarias. Una hélice puede enrollarse con otra y formar una superhélice. También hay combinaciones de hélices y hojas plegadas. Como son muy frecuentes deben ser muy estables.
Estructura terciaria
Mantenida por interacciones de las cadenas laterales. Las cadenas polipeptídicas no se ordenan sólo en estructura de α-hélice porque se quedarían todas las cadenas laterales fuera y muchas son hidrófobas, lo que generaría inestabilidad, por lo que la proteína se pliega sobre sí misma dando lugar a una proteína compacta globular. Este plegamiento es complicado y sin simetría. La norma que rige el plegamiento es que se pliega para esconder los residuos hidrófobos del agua. En una proteína globular la mayoría de los aminoácidos hidrófobos están mirando hacia dentro para quedar excluidos, dejando fuera los residuos polares, por lo que la proteína es soluble. Si quedan residuos hidrófobos hacia fuera la proteína será menos soluble. La excepción son los residuos polares que la proteína guarda dentro para funciones específicas. Si la proteína debe meter muchos residuos hidrófobos protegidos, un trozo de esqueleto polipeptídico se queda mirando hacia dentro, con lo que sus grupos carbonilo y amino están en el interior, pero es más favorable que estén fuera. Esto se soluciona emparejando estos grupos que es lo que se consigue con la α-hélice y la β-hoja. Una proteína se pliega formando el mayor número de puentes de hidrógeno internos dentro de la cadena polipeptídica formados preferentemente entre elementos del enlace peptídico, por lo que la cadena será estable dentro de la proteína. Los grupos que puedan interaccionar con el agua se quedan hacia fuera, por lo que habrán muchas interacciones débiles con el agua participando las cadenas polares. Los elementos del enlace peptídico no interaccionan con el agua sino entre ellos.
Manutención de la estructura terciaria:
- Puentes de hidrógeno e interacciones iónicas entre las cadenas laterales
- Fuerzas de Van der Waals: sólo cuando los residuos están muy cercanos, ya que esta interacción se da entre las partes hidrófobas escondidas dentro de la proteína
- Puentes bisulfuro: únicamente entre las cadenas laterales de cisteínas. De todas las interacciones son las únicas covalentes
Las cadenas laterales que interaccionan pueden estar muy alejadas entre sí en la cadena polipeptídica, cosa que no ocurre en la secundaria. Hay trozos de la proteína sin estructura porque aunque los aminoácidos sean compatibles no puede dar la vuelta y es necesario para la estructura final.
Proteínas en entorno hidrófobo:
Son las proteínas de la membrana que tienen aminoácidos con cadenas laterales hidrófobas, Presentan estructura en α-hélice para que todas las cadenas laterales estén hacia fuera.
Dominios estructurales:
Es un nivel de organización anterior a la estructura terciaria. Al observar una proteína globular plegada se ven distintas regiones bien definidas. Para que haya más de uno la proteína debe ser grande. Los dominios pueden ser iguales entre sí o distintos, se distinguen porque se ve una norma de plegamiento definida. Una proteína con dos dominios tiene dos regiones conectadas por un trozo de cadena polipeptídica (ambas regiones pertenecen a la misma cadena). Un dominio es como una etapa del plegamiento, el primer dominio adquiere su estructura antes de que el segundo esté sintetizado.
Las proteínas pueden tener varias cadenas polipeptídicas, reciben el nombre de oligoméricas y cada cadena es una subunidad.
Estructura cuaternaria
Estudio de cómo se asocian las subunidades para dar la proteína funcional en la que todas las subunidades son importantes. Cada subunidad tiene su estructura terciaria. Las cadenas pueden ser iguales o distintas y a veces unas son funcionales y las otras reguladoras (para regular la actividad de la proteína). Las que mejor pueden regular su actividad son las oligoméricas.
Los grupos que participan en la estructura cuaternaria son los que han quedado hacia fuera. A veces no se esconden todos los grupos hidrofóbicos y así puede asociarse a otra subunidad. Las cadenas no se unen con muchos enlaces bisulfuro para no ser demasiado rígidas. La ventaja de las oligoméricas frente a las monoméricas es que frente a errores de transcripción aunque una subunidad no funcione no se pierde la funcionalidad de la proteína entera, disminuyendo el impacto de los errores.
Ejemplos de proteínas según su estructura tridimensional
Proteínas fibrosas
Aspecto de fibra, formadas por cadenas polipeptídicas paralelas en torno a un eje. Si los residuos son hidrófobos serán insolubles en agua. Su función es estructural, y si fueran solubles se desharían con el tiempo.
Queratina:
Forma parte de la piel y derivados (lana, uñas, pelo …). Según composición y propiedades hay 2 grupos:
1-α-queratina: Tienen mucha cisteína que forma puentes bisulfuro. Cuanta más cisteína hay más dura es la proteína gracias a los enlaces covalentes. La α-queratina tiene un contenido muy alto en cisteína. Si no tiene muchos puentes bisulfuro se puede estirar porque su estructura es una α-hélice con los residuos hidrófobos hacia fuera y los aminoácidos incompatibles con la α-hélice no son frecuentes. Las α-hélices se asocian enrollándose una sobre otra en una estructura supersecundaria, sin plegarse sobre sí mismas. Cada microfibra está formada por 11 microfibrillas, 2 en el centro y 9 rodeándolas. Cada microfibrilla está formada por dos grupos de 2-α-hélices enrolladas hacia la derecha que se asocian enrollándose hacia la izquierda. Estas estructuras son estabilizadas por puentes de hidrógeno y las fibras se mantienen juntas por puentes bisulfuro.
2-Fibroína: Algunas queratinas no tienen cisteína, pero tienen muchos aminoácidos de cadenas pequeñas como glicina, alanina y serina. Una estructura frecuente es que uno de cada dos sea una glicina:
Gly - Ala - Gly - Ser - Gly - Ala …
La Fibroína adopta disposición de hoja plegada asociándose en láminas de hojas. No son extensibles. Las cadenas de aminoácidos son antiparalelas por lo que los puentes de hidrógeno son perpendiculares al eje de avance de la cadena y son más fuertes. Estructura secundaria, las cadenas laterales de los aminoácidos se quedan por arriba y por debajo de la hoja. Como uno de cada dos aminoácidos es glicina (su cadena lateral es un hidrógeno) al apilarse las hojas quedan huecos desiguales entre ellas. Las láminas se unen por interacciones de Van der Waals.
Colágeno:
Su cadena polipeptídica no se dispone ni en α-hélice ni en hoja plegada. Es muy importante cuantitativamente como es el caso de los mamíferos donde llega a ser el 30 % de todas las proteínas. Está presente en todos los organismos pluricelulares. Tiene función estructural formando parte de huesos, tendones, cartílagos … Está formada por fibras paralelas y al microscopio electrónico se ven estrías transversales separadas unos 70 nm.
Composición en aminoácidos: es muy particular, pues ¹/₃ de los aminoácidos son glicina y hay mucha alanina, además de otros raros en gran proporción como prolina y aminoácidos modificados (no codificables) como hidroxiprolina (Hyp) e hidroxilisina (Hyl). El 50 % de las prolinas presentan hidroxilación en el C₃ ó C₄ y las lisinas en el C₅. El OH sirve para establecer puentes de hidrógeno que estabilicen la estructura. También tiene azúcares unidos a la hidroxilisina (monosacáridos y disacáridos). Secuencialmente la glicina es uno de cada tres aminoácidos, lo que es imprescindible para la estructura del colágeno. Como también es rico en hidroxiprolina e hidroxilisina una secuencia típica es:
Gly - Pro - Hyp - Gly - X - Pro - Gly - Hyp - O - …
Estructura tridimensional: La unidad estructural es una fibra de triple hélice enrollada hacia la derecha llamada tropocolágeno. Es una proteína de las más largas y estrechas mide unos 3.000 ángstrom de largo y 15 de ancho. La triple hélice es un elemento de estructura secundaria mantenida por puentes de hidrógeno transversales al eje del tropocolágeno. Es muy estable y resistente. Tiene muchas prolinas e hidroxiprolinas que son aminoácidos de cadenas laterales grandes que rompen la α-hélice. Cada cadena tiene tres residuos por vuelta, estable por repulsiones de tipo estérico y las prolinas están lo más alejadas unas de otras. Cuando se asocian tres cadenas uno de los aminoácidos se tiene que quedar dentro, por eso hay tanta glicina. Se establecen puentes de hidrógeno entre grupos del enlace peptídico. La prolina no tiene dador porque la cadena vuelve a unirse al aminoácido. La cadena polipeptídica tiene cabeza y cola diferenciadas porque la secuencia de aminoácidos no permite adquirir estructura, como consecuencia el principio y el final de la triple hélice está un poco desordenado. Cuando se unen las cadenas se desplazan un poco unas sobre otras y al solaparse cabezas y colas se ven las estrías. Hay dos características que explican su gran estabilidad:
Hay muchos aminoácidos hidroxilados que pueden aportar un OH extra para formar puentes de hidrógeno, sin los aminoácidos hidroxilados el colágeno es más frágil. La hidroxilación de la prolina se produce cuando ésta ya forma parte de la cadena gracias a la enzima hidroxilasa. Si ésta falla el colágeno será más frágil. El escorbuto es una carencia de vitamina C que forma parte de la composición de la hidroxilasa, al escasear produce fragilidad y consiguiente rotura de capilares. Hay enlaces covalentes cruzados entre la misma triple hélice o entre distintas que se establecen entre un residuos de lisina estando una de ellas oxidada por una oxidasa. El número de enlaces covalentes cruzados aumenta con la edad haciendo los huesos más quebradizos.
Proteínas globulares
Al medir volumen de la proteína y compararlo con el tamaño de su cadena polipeptídica se observa que están muy plegadas. Son solubles porque tienen los residuos hidrofóbicos escondidos del entorno acuoso y los polares fuera. Si pierden la estructura terciaria se desnaturalizan y ya no son funcionales ni solubles.
Mioglobina: su estructura fue propuesta por John Cowdery Kendrew. Tiene una propiedad especial que es la capacidad de unir O₂ y almacenarlo de manera reversible. Como no hay ningún aminoácido capaz de unir O₂ a su cadena lateral necesita un grupo prostético. Este grupo es de naturaleza no proteica y forma parte de la proteína para hacerla funcional. El de la mioglobina es un grupo hemo. La mioglobina tiene una alto contenido en α-hélice, hasta 8 tramos. Al plegarse lo hace manera que forma un bolsillo para el grupo hemo cerca de la superficie para que atrape el O₂. Tiene sólo 2 residuos polares (histidina) que en lugar de estar fuera están dentro para unir el grupo hemo. Todos los enlaces peptídicos están en posición trans y es tan compacta que dentro sólo caben 4 moléculas de agua. También tiene prolina para darle la vuelta a la α-hélice. Como existe una relación directa entre la estructura de la proteína y su función las mioglobinas de organismos diferentes son muy parecidas, pero hay aminoácidos que cambian y otros no. No cambian los que son esenciales para la estructura tridimensional, pero hay otros que pueden ser sustituidos por otros de parecidas propiedades. Las dos histidinas son comunes a todas.
Proteínas globulares oligoméricas: la hemoglobina tiene 4 subunidades parecidas a la mioglobina (presenta estructura cuaternaria). Cada subunidad tiene estructura terciaria y se pliegan igual que la mioglobina y tienen un grupo hemo cada una. Para que sea funcional las 4 subunidades han de estar juntas porque si no se quedan con los residuos apolares hacia fuera.
Plegamiento de proteínas
El proceso de plegamiento es secuencial (la estructura se adquiere poco a poco) y cooperativo. Es cooperativo porque cuando se forma un elemento estructural se favorece la formación del siguiente. El esqueleto covalente tiene muchas posibilidades de ordenamiento espacial pero no se pliegan probando estructuras estables sino que cuando prueba a plegarse y encuentra un intermediario apropiado (un trozo bien hecho) se guarda y se va estabilizando por interacciones débiles. La principal directriz de una cadena es esconder los residuos hidrofóbicos. Luego debe adoptar la posición en que mayor número de puentes de hidrógeno pueda formar entre elementos del enlace peptídico. Las cadenas laterales hidrofílicas se dejan fuera para que formen puentes de hidrógeno con el agua. El plegamiento está condicionado por la secuencia de aminoácidos, la estructura se adquiere siempre espontáneamente por lo que es un proceso termodinámicamente favorecido (ΔG < 0). Como ΔG = ΔH - T·ΔS, el factor entrópico se opone al ordenamiento pero gracias a todas las interacciones que se forman el factor entálpico lo compensa. La estructura final será la que más interacciones tenga aparte del enlace peptídico.
Desnaturalización de proteínas
Al modificar el entorno de la proteína podemos hacer que pierda su estructura tridimensional, desnaturalizándola, es decir, desplegando la cadena polipeptídica sin romper los enlaces peptídicos. Ahora la proteína será insoluble y no funcional. Para desnaturalizar una proteína se puede modificar el medio de varias maneras:
- Cambiando el pH cambia el estado de disociación de los grupos y las interacciones no se producirán
- Aumentando la temperatura aumenta la agitación térmica que rompe los enlaces débiles
- Añadiendo sustancias que formen muchos puentes de hidrógeno y compitan con el agua:
![]()
Urea: el NH₂ es dador y el C=O aceptor
Cloruro de guanidíneo:
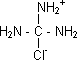
Añadiendo un detergente: La cola hidrofílica se mete dentro de la proteína y las cadenas laterales interaccionan con ella. El SDS, dodecilsulfato sódico, tiene una larga cadena hidrocarbonada y un grupo sulfato cargado.
En el puente bisulfuro los S están oxidados. Dos cisteínas se unen para formar una cistina:
Cys - S -- S - Cys
Estructura terciaria entre cadenas laterales. La proteína no se pliega buscando enlaces bisulfuros sino puentes de hidrógeno. Si al plegarse la proteína parte esconde los residuos hidrofóbicos dos cisteínas caen juntas se formará enlace bisulfuro. Los puentes bisulfuro que tenga una proteína no se deshacen con los métodos anteriores.
Para romperlo:
Reducir el bisulfuro con un reductor. Es reversible. El agente reductor puede ser:
![]()
El mercaptoetanol se oxida y los SH de la proteína se reducen. Al añadir oxígeno se vuelven a formar.
- Ácido perfórmico: el enlace se oxida completamente pasando a SO₃⁻ y la cisteína se convierte en ácido cisteico. No es reversible
- Ácido tricloroacético
Hay que señalar que para romper los puentes bisulfuro hay que añadir antes urea porque pueden estar tan dentro de la molécula que el reactivo no llegue. Para eliminar la urea y el mercatoetanol se usa la diálisis. Esta consiste en la separación de moléculas en base a su tamaño mediante una membrana semipermeable. Se pone la disolución a separar en una bolsa de diálisis y se la rodea de un media sin reactivo, se establece el equilibrio trasvasando las moléculas pequeñas. Se va renovando el medio hasta la total extracción.
Experiencia de Anfinsen: Al principio se pensaba que la desnaturalización era irreversible, Christian Boehmer Anfinsen experimentó con una proteína globular pequeña llamada ribonucleasa (RNasa). Esta proteína tiene unos 120 aminoácidos con 8 cisteínas que pueden formar 4 enlaces disulfuro. Anfinsen añadió urea 8 M y mercatoetanol a la ribonucleasa y la desplegó completamente anulando sus funciones biológicas. Al eliminar la urea y el mercatoetanol se volvieron a formar los puentes de hidrógeno y los bisulfuros, la estructura se rehizo y recuperó el 100 % de su actividad biológica. Las conclusiones fueron:
- El plegamiento es espontáneo, por lo tanto, es termodinámicamente favorable
- La información sobre el plegamiento reside sólo en la cadena de aminoácidos
Hay que tener en cuenta que en un experimento in vitro la concentración de proteínas es pequeña, pero en la célula es muy concentrada y el plegamiento es más eficaz porque hay proteínas que ayudan llamadas carabinas moleculares o chaperones y otras que rompen bisulfuros para que se plieguen bien.
Al poner mercaptoetanol y urea y eliminar ésta después por diálisis se recupera el 1 % de actividad biológica. Esto es porque se forman todos los puentes bisulfuro posibles antes de que adopte su conformación. Si todos los bisulfuros son incorrectos se recuperaría el 0 % de actividad, pero como algunos se establecen correctamente se recupera realmente el 1 %. Para que la proteína sea totalmente funcional se promueve la ruptura de los bisulfuros con mercaptoetanol y se plegará. Luego se elimina.
Editor: Ricardo Santiago Netto (Administrador de Fisicanet).